李亮1)隋志伟1)王晶1)臧超1)余笑波2)
(1)中国计量科学研究院,北京100013 ;2)浙江省计量科学研究院,杭州310013)
摘要 数字PCR是一项针对单分子目标DNA的绝对定量技术。该技术是将含有DNA模板的反应溶液分配到大量独立的反应室中并且发生扩增反应,通过统计反应室中的阳性信号来定量DNA的拷贝数。DNA样品在反应室中随机和独立分布是单分子成功扩增和准确定量D NA拷贝数的关键因素。本文综述了数字PCR的发展历史、数字PCR与实时荧光定量P CR的区别,以及数字PCR在临床诊断、转基因成分定量、单细胞基因表达、环境微生物检测和下一代测序等方面的最新进展,并展望了该技术的应用前景
关键词 数字PCR,绝对定量,实时荧光定量PCR,拷贝数,单分子。
核酸的序列、多样性和丰度分析是现代生物学的基础。核酸定量技术在诊断和个体化医疗、食品检验和转基因生物检测、病原体鉴定、法医科学等方面已被广泛应用,同时这些应用也推动了核酸定量技术的进步。
DNA分子扩增技术的应用促进了分子生物学技术的发展[2]。精确定量 DNA 拷贝数是现代分子生物学和医学的重要应用之一。数字PCR(digital polymerase chain reaction,dPCR)作为 DNA 定量的新技术,克服了实时荧光定量PCR (real timequantitative PCR,qPCR)的一系列缺点,实现了单分子 DNA 绝对定量。dPCR是将单个 DNA 样品反应液分别进行数以百计的反应,并且每个反应分别进行扩增检测。dPCR是 DNA 绝对定量方法,解决了qPCR 所用的标准曲线对测量结果产生影响等问题,并可减少 qPCR 带来的基体效应。此技术在临床诊断、转基因成分定量、单细胞基因表达、环境微生物检测和下一代测序等方面的研究发挥了重要作用。
1 dPCR技术发展历史
1992年,Sykes 等报道了基于样品稀释和泊松分布数据处理的巢式PCR定量技术,并提出了数字PCR的构想。1997年,Kalinina 等使用玻璃毛细管进行了微升(μl) 级的PCR反应,通过荧光探针收集到基因组DNA的单分子信号,进而发展了单分子定量技术。1999年, Vogelstein 和Kinzler基于以上报道采用96孔板系统发展了微升级的PCR扩增方法,用于结肠癌的致癌突变基因ras 的定量分析。随之,Dressman 等发明了一种BEAMing方法( 珠子、乳剂、扩增与磁力):将链霉亲和素与磁珠共价结合,磁珠外包裹一层生物素的PCR引物,进行扩增反应。反乳化作用后,使用流式细胞仪通过磁珠分析等位基因变异。尽管步骤较多,该技术仍不失为广泛应用于实验室定量DNA的理想方法,可以用来检测和定量单核苷酸多态性(single nucleotide polymorphism,SNP) 或突变等核酸序列变异及含有变异等位基因的磁珠能够被流体分离排序,因而可应用于样品测序等序列分析。
尽管dPCR技术具备广阔应用前景,但由于96孔或384 孔平板加样的复杂操作为精确测量带来了困难,也难以解决高通量测量问题。微流体的出现和纳升(nl) 反应仪器的开发克服了这些技术的瓶颈。在2003年,Liu等使用微流体的概念,在微流体芯片上进行了400 个独立的3nl PCR反应。2008 年,嵌入式芯片的PCR 反应次数达到了9180 个6nl 的PCR平行反应。例如,Fluidigm公司的Biomark 系统,此系统微流体dPCR芯片由12个独立的微流体面板(panel) 和相应进样孔组成(图1). 每个面板含有765 个6nl 的独立反应室(partition)。4.6μl反应液通过进样孔进入后被分配到765个反应室进行独立扩增。该技术在日趋完善的同时,单次的反应数和检测精度成为此项技术研究的重点。最近,Heyries 等报道了一个百万级的微流体dPCR装置,成为了dPCR技术的又一重大突破。该装置通过表面张力以达到样品分布和疏水控制,提高了单分子DNA扩增的保真性,每皮升( pl) 进行100万个反应,达到每平方厘米(cm2) 44万个反应。这种装置可实现每10万个野生型序列中低于一个变异拷贝的检测。

图1 微流体dPCR的12×765 反应芯片
2 qPCR与dPCR比较研究
qPCR是目前DNA定量研究的主要技术,该技术通过在PCR反应体系中加入荧光结合染料(SYBR green I) 或荧光标记的探针( 如TaqMan Probes) ,利用实时积累的荧光信号监测整个扩增过程,最后通过标准曲线对未知模板进行定量分析,以此来评估PCR的扩增效果。qPCR主要依赖于校准物制备的标准曲线,进而确定未知样品的浓度,因此是一种相对定量的方法。该技术存在以下问题:a、校准品和样品间背景的不同将引入偏差,并且影响PCR的效率和测量响应;b、低拷贝数的目标 DNA分子不能通过扩增检测到;c、样品的PCR扩增效率可能与校准物的扩增效率不同;d、DNA提取时引入DNA溶液的杂质或者DNA降解影响了PCR动态扩增过程。
dPCR是一项检测和定量核酸的新技术。它不同于传统的qPCR,因为采用直接计数目标分子数而不依靠任何校准物或外标,dPCR通过计数单个分子从而实现绝对定量。dPCR将传统PCR的指数数据转换成数字信号,仅仅通过显示程序设定的循环数后扩增是否发生,即可克服上述困难,达到核酸的绝对定量。dPCR在进行扩增反应前,将含有DNA模板的PCR溶液稀释后分布到大量的独立反应室(图2) ,单分子间通过稀释分离,并且独自进行PCR扩增,最后分析每个扩增产物。这实现了通过先于PCR扩增的样品分离。这种样品的分配可以消除本底信号的影响,提高低 丰度靶标的扩增灵敏度,简单计算出DNA的模板拷贝数而不需要采用参考标准物或者外标。准确的定量依赖于40~45个循环的扩增,错误的阴性检出水平(单DNA模板出现在反应室而未被检出) 非常低。数字PCR是一项具有可重复性的定量微量DNA分子的优良技术。其操作方便、检测通量高、特异性强、灵敏度高、定量准确等优点使其成为了分子生物学研究中的重要工具。
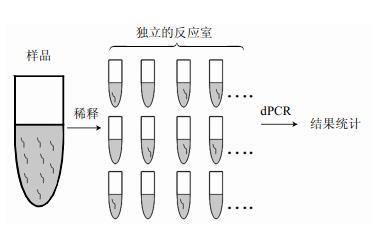
图2 dPCR扩增反应原理
3 dPCR应用研究
3.1 dPCR在临床诊断方面的研究
dPCR应用于癌症的等位基因突变和拷贝数变异等方面的检测研究,为癌症检测提供了新的诊断工具。Oehler 等对慢性白血病的相关基因ABL酪氨酸激酶结构域突变采用了dPCR进行绝对定量检测,并且建立了同时检测多个ABL酪氨酸激酶结构域突变的方法。表皮生长因子受体( epithelial growth factor receptor ,EGFR) 突变或过表达一般会引发肿瘤。Yung 等开发了一种关于非小细胞性肺癌患者血浆和肿瘤中的两种EGFR突变体( 第19外显子内缺失和第21外显子L858R 突变) 的dPCR定量检测方法。第19外显子内缺失和第21外显子L858R突变占酪氨酸激酶突变响应的85% 以上。实验结果表明这两种突变在预治疗的35个血浆样品分别检出6个(17%)和9个(26%)。与肿瘤细胞的测序结果相比,血浆EGFR突变的灵敏度和特异性是92% 和100%。在肿瘤和血浆中的低丰度EGFR突变通过微流体dPCR灵敏检测和精确定量将应用于预测治疗反应、监控疾病进展和早期检测获得性耐药等方面研究。
高通量基因分型验证与疾病相关的SNPs研究促使研究者采用一项新的技术,dPCR,对外周血液及口腔洗液提取DNA的基因分型进行评价分析。利用基于BioMark PCR平台的Fluidigm 48×48动态阵列生物芯片进行SNP基因分型。共90个样品与20个SNP的体系对比,分别得到平均99.7%以及16个体系100% 的结果。TaqMan 基因分型与三组SNP进行对比得到了100%的相关性。为了研究DNA模板变化的影响,血液样品( CH-1,n=20;CH-2,n=47;KK,n=47)和口腔洗液( n=37) 通过24个SNPs 进行基因分型。CH-1 和CH-2 批次显示了很好的基本响应(≥98.8%),KK批次和口腔洗液样品响应较低(82.1% 和94.0 %)。但经再纯化后的KK和口腔洗液样品结果响应率有显著提高( 逸98.8% ).研究结果表明dPCR的准确度与TaqMan 基因分型类似,但DNA用量更少,节省了更多的人力、时间及成本。
拷贝数变异(copy number variation,CNV) 是人类遗传变异的重要来源,并且与大量的人类疾病密切相关。Qin等采用dPCR 系统精确定量了DNA样品的拷贝数,建立了dPCR的分析模型,并且进行了以下3个方面的研究:a、在一系列人类基因组和合成的RPP30基因中定量了RPP30基因的拷贝数,与预期结果相符;b、研究了商品化样品中的CYP2D6基因拷贝数,与传统手段定义的拷贝数一致;c、通过对ERBB2 基因的扩增筛选到了40个乳腺癌样品,与文献报道的基本一致。这项研究表明,dPCR为特异性CNV 的研究供了一个新的、精确的和强有力的技术支持。
孕妇血浆中的胎儿游离核酸(DNA和RNA检测开启了无创产前诊断的新领域,补充了唐氏综合症(非侵入性染色体异倍体) 产前诊方法。基于胎儿核酸的精确dPCR方法推进了此研究领域的发展,为孕妇及胎儿提供更安全诊断。在2007年,Lo等在证明了dPCR 可用于孕妇血浆RNA- SNP等位基因比率的测量后,应用该方法从整倍体胎盘DNA 样品中鉴别出21个三倍体样品。Lo等进一步证明:即使三倍体DNA以小量片段存在时,异倍体也能被检测到。后者对于针对母亲血浆建立的三倍体检测方式是十分必要的。Fan和Quake后续发表的文献也支持了这个观点。随后,Lo等针对以蛋白质- RNA 复合物方式存在于血浆中的胎儿游离RNA进行了研究。比较了质谱和dPCR对孕妇血浆中PLAC4基因的mRNA-SNP比率,其中质谱的临床灵敏度为90%,临床特异性为96.5%,dPCR的临床灵敏度和特异性均为100%。近期研究表明,与质谱检测和qPCR相比,dPCR可以获得更精确的定量结果。故此,dPCR检测胎儿DNA的浓度可能要高于预期。微流体芯片dPCR的快速发展,已成为目前临床应用最具潜力的无创产前诊断技术,并为其在常规检查中的应用奠定了基础。
3.2 dPCR在转基因植物检测方面的研究
转基因植物及相关食品的定量分析主要测定转入基因的相对含量。目前常用qPCR作为核酸定量方法。dPCR可以不需要校准物而准确测量低拷贝的DNA分子。Corbisier 等用dPCR分析了提取于MON810玉米种子的外源检测基因和hmg基因的拷贝数,验证了dPCR的绝对拷贝数比例定量结果和质粒DNA做标准物质qPCR相对定量比率一致。因此提出dPCR具有计量特性,可以用来测量转基因相关标准物质的D NA拷贝数比率。
单分子扩增效率对于改善总DNA片段的拷贝数和短片段完整DNA的估计偏差有显著意义。目标DNA分子在反应室的随机和独立分布是dPCR准确定量的关键。Bhat 等认为,反应室的容量是不确定度的主要来源,并且评定的相对不确定度在6%以下。这项发现可以用于其他dPCR测量的置信水平研究。随后,Burns 等采用经验证的检测转基因成分的qPCR反应体系,评估了dPCR技术的绝对检测限和定量限,也阐述了采用梯度预实验的方式可以使dPCR更精确地测量拷贝数。经过30个平板重复后,实验结果表明在每个平板发生200 ~700个反应是最为精确的,这和仪器厂商的推荐是一致的。
3.3 dP CR在单细胞基因表达方面的研究
将dPCR 技术用于单细胞基因表达研究是dPCR技术发展的里程碑。White 等设计开发了一种能够高精度测量单细胞中数以百计的基因表达的dPCR设备。此设备可进行包括细胞获取、裂解、反转录和定量PCR等单细胞加工处理。此外,为了提高通量,降低成本,White 等采用微升体积处理减少了测量噪音,增加了灵敏度,提高了单核苷酸的特异性。并且应用这项技术测定了3300个单细胞,包括:a、miRNA在K562 细胞的表达;b、miRNA及其目标转录子在胚胎细胞分化时的协同作用;c、乳腺癌细胞的单核苷突变检测等,建立的核心功能提供了多种基于芯片系统的单细胞转录分析。
Spurgeon等报道了基于微流体dPCR的高通量基因表达平台。此平台同时对2304个基因进行扩增反应,与96孔板相比需要更少的上样量。在单芯片上还可以检测18个组织中的45个不同基因。实验结果与传统的qPCR十分吻合,并且同商业化的DNA芯片相比具有更好的重复性。Warren等报道了数字RT-PCR微流体芯片可实现转录因子表达数量的系统定量分析,并且可以计算源自单细胞的cDNA样品。在一个包括早期5 级造血前体的研究中,Warren 发现造血干细胞中转录因子PU1表达显著变化,在flk2-和flk2+骨髓祖细胞中PU1表达呈多样性。
最近,Guo等采用dPCR技术研究了受精卵到囊胚的单细胞基因表达。利用单细胞表达分析,对小鼠囊胚中64细胞期3 种不同细胞类型的500个细胞分化到此阶段进行了研究。基于800余个转录子的全胚胎分析,选择了48个基因进行研究,结果发现3 种细胞类型可以利用定量表达方法进行明显的区分。最后分别确定ID2和Sox2为细胞外和细胞内最早的标记物。进而对早期内细胞受体配合对Fgfr2/Fgf4 进行负相关表达分析。定位与信号传递发生在转录活动成熟之前。结果表明单细胞表达分析能够深入研究细胞的发育机制,应将这项技术推广应用于更广泛的生物系统中。
3.4 dP CR在环境微生物方向的研究
dPCR技术高通量扩增和分析的优点使同时研究自然界的多个细菌单细胞的多个不同基因得以实现。Ottesen 等使用一个在白蚁和其肠道菌群相关的多级关键酶基因作为诱饵,采用dPCR技术发现了未知的核糖体RNA。这种技术能够系统地鉴定复杂生态环境中携带目的基因的细菌,并根据一两个目的基因追溯到其种属,因此,dPCR将为环境研究领域提供新的契机。
病毒可能是地球上存在量最大的生物物种。然而,对于大多数病毒的寄主,却鲜有基因组学或经典的阶段划分技术对其进行研究。Tadmor 等采用dPCR方法,通过一个病毒标志基因将单细菌细胞与自然环境相关联。应用这项技术对白蚁后肠微生物群落的研究发现在属的范围感染模式间,存在着巨大的属内选择性。病毒标志物显示了宿主间严格的等位基因混合,揭示了除邻近宿主外的等位基因横向基因转移的限制程度。方法无需培养宿主细胞或病毒,为许多环境细菌与病毒的相互作用提供了有效方法。
3.5 基于dPCR的单分子测序技术研究
自从2003年单分子测序技术应用到测序领域后,此类技术的各类形式相继报道,并且以每年10倍通量高速增长。
dPCR为高通量测序提供了灵敏的和绝对的校准。下一代测序(next generation sequencing, NGS)如454,Solexa 和SOLiD平台需要通过测序校正分子数量。此条件存在两种不利后果:a、大量微克级的样品需要制备为文库,因此限制了可测序样品的范围。对多数应用来讲,包括宏基因组、考古学样品、法医样品和临床样品测序,DNA的样品量是非常有限的。b、每个文库需要滴定法测序,因此增加了成本, 降低了测序的通量. 为此, 使用dPCR精确定量454 和Solexa 测序文库,使测序文库的制备达到纳克级,同时消除了花费在仪器滴定的成本和时间。同时成功地对454 FLX 和Sole xa测序平台的低于纳克级的细菌和哺乳动物的DNA样品进行了测序。White 等的研究结果首次明确证明了基于454 平台的皮克级DNA样品的测序,对DNA样品的需求量在无预扩增时降低了1000 余倍。dPCR实现了测序文库的绝对定量,消除了构建和PCR定量标准曲线等不确定因素,相对标准偏差低于10% ,在无滴定情况下,足够满足直接测序的精度要求。
ABCA4基因突变或等位基因不一致可引起多种疾病。Zernant 等为寻找ABCA4疾病的相关变异基因,研究了168 例医学诊断为黄斑病变、视锥-视杆营养不良或其他ABCA4疾病表型的患者。首先使用ABCA4芯片筛选基因突变,结果发现111 例病人中有1 例病人有2 个预期的突变,57个病人中没有,随后应用dPCR和454 测序平台鉴定了新变异结果,分析了这些致病性的原因。在已鉴定单一位点突变的103 例病人中,鉴定了第二疾病相关等位基因49例。虽然编码 ABCA4基因的众多突变仍然未知,并且许多位于ABCA4的非编码区,但是ABCA4实验结果是一个较好的选择方法,NGS 平台对于筛选大量的疾病是一个在时间和消费方面都高效的工具。
NGS 是一种识别和确认未知致病菌的前景广阔的技术,然而其在生物防御和公共健康应用等方面的时效性,却往往因为缺乏快速、有效、可靠的自动DNA样品制备方法而受到限制。为了突破这种限制,Kim 等设计了一种基于流体分布元件的数字微流体(DMF) 平台,使得多子系统模块能够进入自动NGS库样品制备系统。通过一种新型毛细管接口,可以实现液体在DMF设备与外部流体模块间的高重复性转移,使得流路连续,且小滴样品能够在一个完整的系统内得到处理。这里,该技术强调了DMF元件平台和NGS 样品制备流程中自动运行毛细管接口的效用。将毛细管接口与一种嵌入式非接触电导检测器连接,DMF设备实现了目标分析物从样品流到小液滴的闭环自动片段采集。NGS 中重复次数最多的缓冲液交换与样品清洗通过使用一种磁珠解决,实现了DMF平台上平均DNA回收率为(80±4.8)%。
先天性糖基化缺陷是由于机体缺少糖基化作用,主要影响到N 相关途径造成的。至少40% 的先天性糖基化缺陷病人在诊断过程中没有得到分子水平的确认。Jones 等通过已经导致先天性糖基化缺陷基因的下一代测序验证的研究,从分子水平提高了诊断先天性糖基化缺陷的水平。12例未知样本致病突变的先天性糖基化缺陷病人作为阳性对照进行NGS验证,分别采用RainDance 与Fluidigm 平台( dPCR) 进行序列富集和目标扩增,SOLiD平台用于测序和扩增产物。进而通过NextGENe 进行生物信息学分析。结果表明,12个阳性对照的致病突变通过NGS都得以确认。在病人诊断过程中,NGS的发展使实验室诊断多基因与分析逐个基因相比成本消耗更低、速度更快、效率更高。临床实验室的下一代测序数据分析结果同样支持这项技术,推广使用这项技术至关重要。
4 总结与展望
生物学的基础研究和分子技术的前进伴随着更精确和更灵敏的测量技术发展。值得一提的是,dPCR具有测量独立性与无需任何校准物的特点。因此,该技术是潜在的核酸测量基准方法,并从原理上为核酸计量提供了保证。相比其他方法,dPCR作为绝对定量方法能准确定量目标DNA和提供可靠的定量数据。商业化dPCR 仪器( 如Fluidigm 公司的BioMark System)的大量出现进一步推动和扩大了该技术的发展和应用范围。
dPCR技术及其应用凸显了单分子定量技术的潜力。不久,微流体dPCR就会突破反应速度和体积的限制,实现自动化和高通量的应用。核酸测序将是基于dPCR的单分子扩增技术最重要的应用领域。dPCR的克隆扩增可以减少下一代测序的时间和成本,并使个人基因组测序得以实现。我们期望在不远的将来,这项技术的发展将对单分子核酸扩增领域产生深远影响,在分子生物学和医学等基础研究和应用方面发挥更大的作用。
参考文献
[1] Keer J T , Birch L. Essentials of Nucleic Acid Analysis: A Robust Approach. London: Royal Society of Chemistry, 2008
[2] Zhang C, Xing D. Single-molecule DNA amplification and analysis using microfluidics.Chem Rev, 2010, 110 (8): 4910-4947
[3] Vogelstein B, Kinzler K W. Digital PCR. Proc Natl Acad Sci USA,1999,96(16): 9236-9241
[4] Bhat S, Herrmann J, Armishaw P, et al. Single molecule detection in nanofluidic digital array enables accurate measurement of DNA copy number. Anal Bioanal Chem, 2009, 394(2): 457-467
[5] Bustin S A, Benes V, Garson J A, et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clin Chem , 2009, 55 (4 ): 611-622
[6] Burns M, Burrell A, Foy C. The applicability of digital PCR for the assessment of detection limits in GMO analysis. European Food Research and Technology, 2010, 231 (3 ): 353-362
[7] Sykes P J , Neoh S H , Brisco M J, et al. Quantitation of targets for PCR by use of limiting dilution. Biotechniques, 1992,13(3): 444
[8] Olga K, Irina L, James B, et al. Nanoliter scale PCR with TaqMan detection. Nucleic Acids Res, 1997,25 (10 ): 1999-2004
[9] Devin D, Hai Y, Giovanni T, et al. Transforming single DNA molecules into fluorescent magnetic particles for detection and enumeration of genetic variations . Proc Natl Acad Sci USA, 2003,100 (15): 8817-8822
[10] Diehl F, Li M, Dressman D, et al. Detection and quantification of mutations in the plasma of patients with colorectal tumors. Proc Natl Acad Sci USA, 2005, 102 (45): 16368-16373
[11] Frank D, Meng L, Yiping H, et al. BEA Ming: single-molecule PCR on micro particles in water-in-oil emulsions. Nat Methods, 2006, 3 (7): 551-559
[12] Liu J , Hansen C, Quake S R. Solving the " world-to-chip" in terface problem with a microfluidic matrix. Anal Chem,2003, 75(18): 4718-4723
[13] Dube S, Qin J , Ramakrishnan R. Mathematic alanalysis of copy number variation in a DNA sample using digital PCR on a nanofluidic device. PLoS One , 2008, 3 (8): e2876
[14] Heyries K A, Carolina T, Michael C, et al. Megapixel digital PCR. Nat Methods, 2011,8 (8): 649-651
[15] Sanders R, Jim F, Huggett, et al. Evaluation of digital PCR for absolute DNA quantification. Anal Chem, 2011, 83(17): 6474-6484
[16] Heid C A, Stevens J, Livak K L, e t al. Real time quantitative PCR. Genome Res, 1996, 6 (10): 986-994
[17] Morrison T B, Weis J J, Wittwer C T. Quantification of low-copy transcripts by continuous SYBR Green I monitoring during amplification. Biotechniques, 1998, 24(6): 954-958 , 960 , 962
[18] 赵锦荣, 白玉杰, 王胜春, 等.新型MGB探针在沙眼衣原体实时PCR检测中的应用. 生物化学与生物物理进展, 2003, 30(3): 466– 470 Zhao J R, Bai Y J , Wang S C, et al. Prog Biochem Biophys, 2003,30(3): 466-470
[19] Corbisier P, Somanath B, Lina P, et al. Absolute quantification of genetically modified MO N810 maize(Zea mays L.) by digital polymerase chain reaction.Anal Bioanal Chem, 2009, 396 (6):2143-2150
[20] Terry C F, Shanahan D J , Ballam L D, et al. Real-time detection of genetically modified soya using Lightcycler and ABI 7700 platforms with TaqMan , Scorpion, and SYBR GreenⅠ chemistries. J AOAC international, 2002, 85 (4): 938- 944
[21] Pohl G, Shih I M. Principle and applications of digital PCR. Expert Re v Mo l Di agn , 2 00 4, 4(1 ): 41- 47
[22] Oehler V G, Qin J, Ramakrishnan R, et al. Absolute quantitative detection of ABL tyrosine kinase domain point mutations in chronic myeloid leukemia using a novel nanofluidic platform and mutation-specific PCR. Leukemia, 2008, 23(2): 396-399
[23] Chan M, Mei W C, Ting W L, et al . Evaluation of nanofluidics technology for high-through put SNP genotyping in a clinical setting. J Mol Diagn, 2011, 13 (3): 305-312
[24] Ropers H H. New perspectives for the e lucidation of genetic disorders . Am J Hum Genet,2007, 81(2 ): 199-207
[25] Lupski J R. Genomic rearrangements and sporadic disease. Nat
Genet, 2007, 39: S43-S47
[26] Slamon D J, Clark G M , Wong S G, et al . Human breast cancer:correlation of relapse and survival with amplification of the HER-2 /neu oncogene .Science, 1987, 235 (4785): 177
[27] Qin J, Jones R C, Ramakrishnan R. Studying copy number variations using a nanofluidic platform. Nucleic Acids Res, 2008,36 (18): e116
[28] Lo Y M D. Noninvasive prenatal detection of fetal chromosomal aneuploidies by maternal plasman ucleic acid analysis: a review of the current state of the art. BJOG: An International J Obstetrics&Gynaecology, 2009, 116 (2): 152-157
[29] Lo Y M D, Fiona M F, Chan K C A, et al. Digital PCR for the molecular detection of fetal chromosomal aneuploidy. Proc Natl Acad Sci USA, 2007, 104(32): 13116 -13121
[30] Fan H C, Quake S R. Detection of aneuploidy with digital polymerase chain reaction. Anal Chem, 2007, 79 (19): 7576-7579
[31] Lo Y M, Nancy B Y T , Rossa W K C, et al. Plasma placental RNA allelic ratio permits noninvasive prenatal chromosomal aneuploidy detection. Nat Med , 2007, 13 (2): 218-223
[32] Lun F M, Allen C K, Yeung L T, et al. Microfluidics digital PCR reveals a higher than expected fraction of fetal DNA in maternal plasma. Clin Chem, 2008, 54(10): 1664-1672
[33] 郭奇伟,周裕林.唐氏综合征的无创产前诊断研究进展. 中国优生与遗传杂志, 2010,18(4): 137-139
Guo Q W, Zhou Y L. Chin J Birth Health & Hered, 2010, 18(4):137-139
[34] BioMark. BioMark Advanced Development Protocol 10. Absolute quantitation using the digital array; Fluidigm (Fluidigm Corporation, S.F)
[35] White A K, Michael V,Oleh I P, et al. High-through put micro fluidic single-cell RT-qPCR. Proc Natl Acad Sci USA, 2011,108(34): 13999-14004
[36] Spurgeon S L, Jones R C, Ramakrishnan R. High throughput gene expression measurement with real time PCR in a microfluidic dynamic array. PLoS One , 2008, 3(2): e1662
[37]Warren L,David B, Irving L W ,et al.Transcription factor profiling in individual hematopoietic progenitors by digital RT-PCR. Proc Natl Acad Sci USA , 2006,103(47):17807-1 7812
[38] Guo G , Huss M, Tong G Q, et al. Resolution of cell fated ecisions revealed by single-cell gene expression an alysis from zygote to blastocyst. Dev Cell , 2011,18 (4): 675-685
[39] Ottesen E A , Jong W H, Stephen R Q, et al. Microfluidic digital PCR enables multigene analysis of individual environmental bacteria.Science ,2006,314 (5804): 1464-1467
[40] Tadmor A D, Elizabeth A O , Jared R L, et al. Probing in dividual environmental bacteria for viruses by using microfluidic digital PCR. Science, 2011,333(6038): 58-62
[41] Braslavsky I, Hebert B, Kartalov E, et al. Sequence information can be obtained from single DNA molecules. Proc Natl Acad Sci USA,2003, 100 (7): 3960-39 64
[42] Eid J, Fehr A, Gray J, et al. Real-time DN A sequencing from single polymerase molecules . Science, 2009, 323 (5910): 133-138
[43] Harris T D, Buzby P R, Babcock H, et al. Single-molecule DNA sequencing of a viral genome. Science, 2008, 320(5872 ): 106 -109
[44] White R A , Blainey P C, Fan H C, et al . Digital PCR provides sensitive and absolute calibration for high throughput sequencing.BMC Genomics, 2009, 10:116
[45] Zernant J , Schubert C, Im K, et al. Analysis of the ABCA4 gene by next-generation sequencing . Invest Ophthalmol Vis Sci, 2011,52(11): 8479-8487
[46] Kim H , Bartsch M S, Renzi R F, et al. Automated digital microfluidic sample preparation for next-generation DNA sequencing. J Lab Autom , 2011, 16(6): 405-414
[47] Jones M A, Bhide S, Chin E, et al. Targeted polymerase chain reaction-based enrichment and next generation sequencing for diagnostic testing of congenital disorders of glycosylation. Genet Med, 2011,13 (11): 921-932